polysome profil
ing 富集检测 ¥6800
广州卿泽生物科技有限公司
入驻年限:5 年
- 联系人:
吴小姐
- 所在地区:
广东 广州市 黄埔区
- 业务范围:
医疗器械、体外诊断、论文服务、细胞库 / 细胞培养、试剂、技术服务
- 经营模式:
经销商 生产厂商
推荐产品
公司新闻/正文
PNAS | HRAS m6A修饰促进肿瘤发展
人阅读 发布时间:2024-04-24 14:49
导读:
RAS基因是第一个被鉴定的人类致癌基因,该基因家族包括3种功能型基因:HRAS、NRAS和KRAS。既往针对Ras的研究主要聚焦于其基因突变或蛋白稳定性调控肿瘤发生和转移的分子机制。转录后修饰是表观遗传机制的重要环节,m6A修饰是真核生物RNA最主要的转录后修饰,其功能失调与包括癌症在内的多种疾病密切相关。RAS基因家族是否具有m6A修饰位点,以及m6A修饰如何调控RAS基因的肿瘤驱动功能尚未见报道。

本研究发现:癌组织中H-Ras(非KRAS和NRAS)普遍存在n6 -甲基腺苷(m6A)修饰,从而导致其蛋白表达增加,以促进癌细胞的增殖和转移。机制上,HRAS的3'UTR区存在3个m6A修饰位点,提高翻译延伸率促进其蛋白表达。这些m6A修饰位点主要受去甲基化酶FTO调控,并可被YTHDF1蛋白识别。靶向HRAS m6A修饰可减少肿瘤的增殖和转移。临床上,在多种癌症中,H-Ras表达上调与FTO表达下调、YTHDF1表达上调相关。
该研究揭示了HRAS特异性m6A修饰位点与肿瘤进展之间的联系,这为靶向致癌Ras信号通路提供了一种新的策略。
文章索引
标题:Epitranscriptic regulation of HRAS by N6-methyladenosine drives tumor progression
发表期刊:Proc Natl Acad Sci U S A.
发表时间:2023.04
作者及单位:东南大学高山教授团队
IF:11.1
DOI:10.1073/pnas.2302291120.
研究思路

研究结果
(1)HRAS m6A修饰鉴定
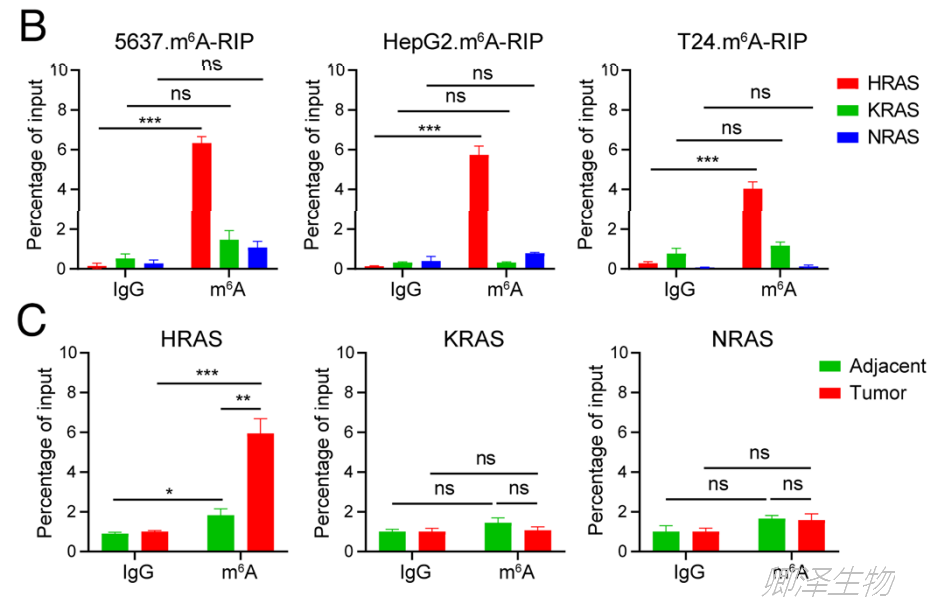
与癌旁组织相比,HRAS的3'UTR在肿瘤组织中具有更高的m6A水平,而5'UTR和CDS没有明显的m6A修饰(图1A)。然而,在KRAS和NRAS中没有观察到明显的m6A修饰(图1A)。

m6A-RIP-qPCR分析发现:膀胱癌5637细胞系、肝癌HepG2细胞系(HRAS野生型)和膀胱癌T24细胞系(HRASG12V突变)的HRAS被m6A显著修饰(图1B)。此外,肾细胞癌组织中HRAS的m6A水平明显高于邻近组织(图1C)。
总之,这些数据表明,m6A修饰存在于HRAS中,而不存在于KRAS和NRAS中。
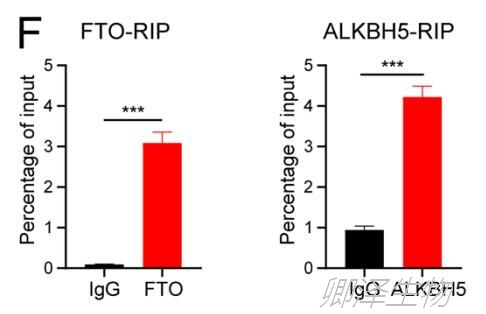
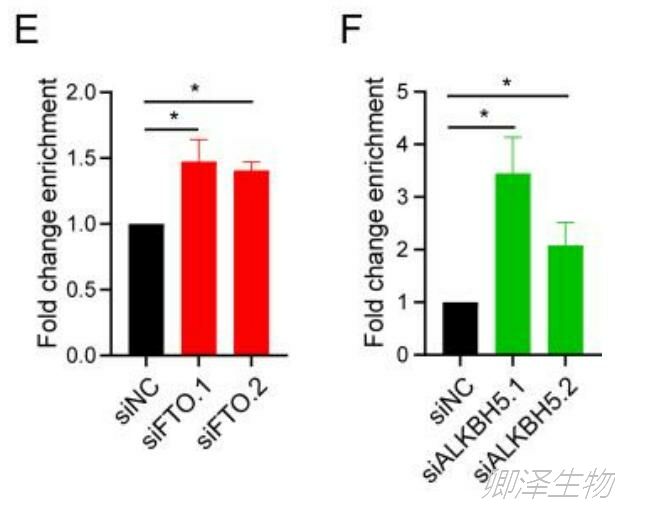
接着,确定HRAS m6A修饰的主要调控因子。m6A-RIP-qPCR分析显示,去甲基化酶FTO和ALKBH5敲低后,HRAS m6A修饰增强(图1D);野生型FTO和ALKBH5过表达,HRAS m6A修饰减弱(图1E)。

此外,RIP-qPCR显示FTO和ALKBH5与HRAS结合(图1F)。总之,这些结果证实了HRAS是FTO和ALKBH5催化的去甲基化的直接底物。
(2)m6A修饰可增强HRAS翻译
m6A修饰对HRAS表达存在哪些影响呢?
首先,荧光素酶报告基因(下图A)、亚细胞定位实验(下图B-C)和HXD处理(下图D-E)排除了m6A对HRAS转录、亚细胞定位、mRNA稳定性的调节。
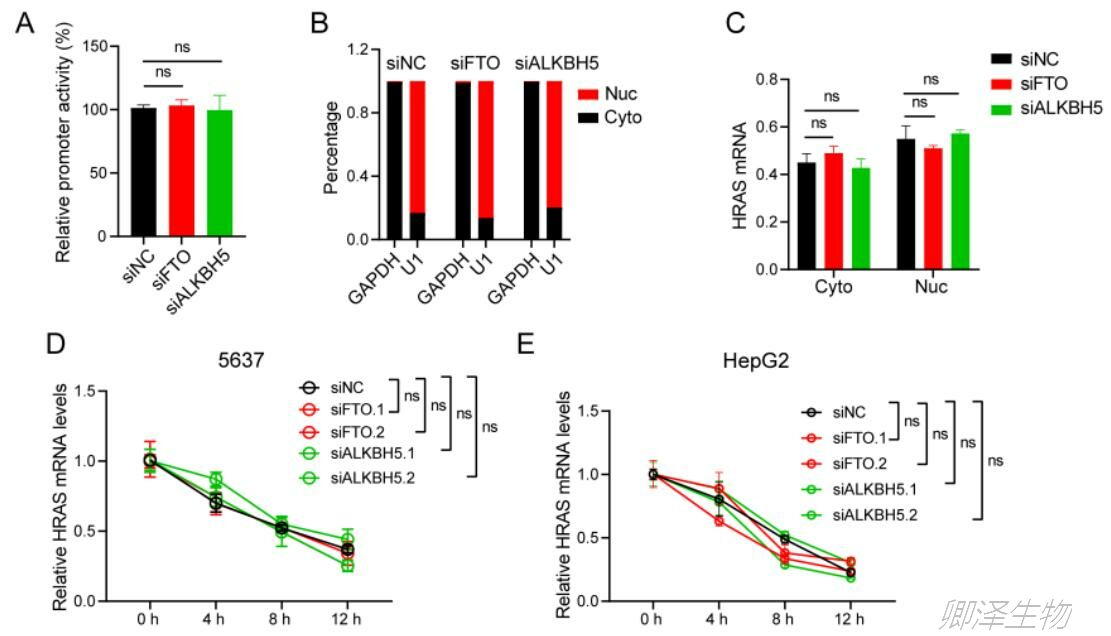
qRT-PCR结果显示,FTO和ALKBH5 KD对HRAS、KRAS和NRAS的mRNA表达均无显著影响(图2A)。这些结果表明m6A对HRAS的RNA水平没有影响。

然而,FTO和ALKBH5 KD均显著增加了H-Ras蛋白水平(图2C-D)。相比之下,野生型FTO和ALKBH5的OE显著降低了H-Ras蛋白水平(图2E-F)。这些结果表明m6A可提高H-Ras蛋白水平。

这怎么回事??翻译组学来解释~~~
首先构建了pmirGLO-HRAS 3'UTR荧光素酶报告基因,探讨m6A是否调控HRAS的翻译。双荧光素酶实验显示,FTO和ALKBH5 KD后,荧光素酶活性显著增强,而萤火虫荧光素酶(F-Luc)和Renilla荧光素酶(R-Luc) mRNA丰度不受影响(图2H),表明去甲基酶调节了HRAS的翻译效率。

接下来,作者用两种方法进行核糖体捕获(23,24)。Polysome profiling及qRT-PCR显示,FTO和ALKBH5 KD后,HRAS主要分布在Polysome组分中(图2I)。

另外,RiboLace显示FTO和ALKBH5 KD后HRAS mRNA分布在活性核糖体结合的片段中。总之,这些数据表明m6A修饰与HRAS的翻译有关。
(3)YTHDF1以依赖m6a的方式促进HRAS翻译
RIP-qPCR检测显示,在5637和HepG2细胞中,YTHDF1与HRAS结合明显(图3A)。RIP-seq显示YTHDF1在HRAS的3'UTR中显著富集(图3B),证实了HRAS的3'UTR被YTHDF1特异性结合。

RIP- qPCR发现FTO和ALKBH5 KD后,YTHDF1与HRAS转录物的结合能力显著增强(图3C)。将YTHDF1的两个关键氨基酸突变(K395A和Y397A),以消除其与m6A的结合。Flag RIP-qPCR检测显示,HRAS与YTHDF1-wt的相互作用显著增强,但YTHDF1-mut与HRAS的相互作用明显减弱(图3D),提示YTHDF1与HRAS转录物的结合依赖于m6A。


Polysome profiling和RiboLace显示:YTHDF1 KD后,Polysome组分或翻译活性核糖体部分中的HRAS mRNA水平降低(图3F和G)。

YTHDF1促进m6A修饰基因的翻译,通常与翻译起始因子和延伸因子相关,如eIF3、eIF4、eEF1和eEF2。RIP-qPCR检测显示eEF1和eIF4与HRAS转录本显著结合(图3I)。FTO和ALKBH5 KD中eEF1对HRAS转录本的结合能力增强(图3 J和K),证实m6A通过YTHDF1和eEF1诱导HRAS mRNA的翻译延伸。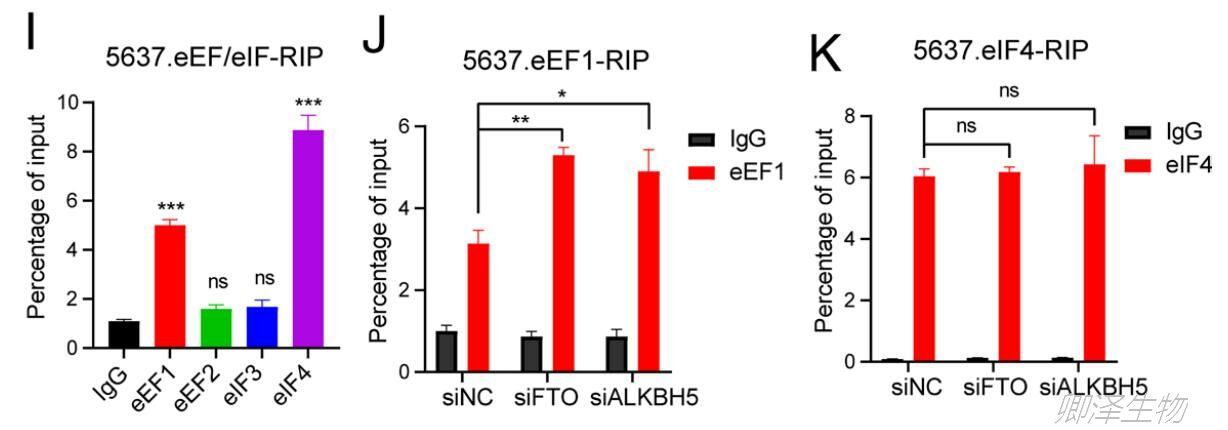 综上所述,这些数据表明YTHDF1以依赖于m6A的方式促进了HRAS的翻译效率。
综上所述,这些数据表明YTHDF1以依赖于m6A的方式促进了HRAS的翻译效率。
生物素化和m6A修饰的RNA探针pull-down实验进一步验证了YTHDF1与三个m6A位点的结合(图4E)。
最后,再综合m6A位点点突变的荧光素酶报告基因(图4F),以及HRAS-Mut和YTHDF1-Mut的IB分析(图4G)结果,表明HRAS 3' UTR包含三个m6A位点,这是YTHDF1结合和HRAS翻译所必需的。




研究结论
该研究解析了HRAS的表观转录调控在驱动肿瘤发生发展中的重要作用与机制,为靶向Ras的肿瘤精准治疗提供了新的理论基础和策略。
欢迎致电了解详情或咨询!
广州卿泽生物科技有限公司
地址:广州市黄埔区伴河路96号一栋三层321房
电话:18925086102(微信同号)
RAS基因是第一个被鉴定的人类致癌基因,该基因家族包括3种功能型基因:HRAS、NRAS和KRAS。既往针对Ras的研究主要聚焦于其基因突变或蛋白稳定性调控肿瘤发生和转移的分子机制。转录后修饰是表观遗传机制的重要环节,m6A修饰是真核生物RNA最主要的转录后修饰,其功能失调与包括癌症在内的多种疾病密切相关。RAS基因家族是否具有m6A修饰位点,以及m6A修饰如何调控RAS基因的肿瘤驱动功能尚未见报道。

本研究发现:癌组织中H-Ras(非KRAS和NRAS)普遍存在n6 -甲基腺苷(m6A)修饰,从而导致其蛋白表达增加,以促进癌细胞的增殖和转移。机制上,HRAS的3'UTR区存在3个m6A修饰位点,提高翻译延伸率促进其蛋白表达。这些m6A修饰位点主要受去甲基化酶FTO调控,并可被YTHDF1蛋白识别。靶向HRAS m6A修饰可减少肿瘤的增殖和转移。临床上,在多种癌症中,H-Ras表达上调与FTO表达下调、YTHDF1表达上调相关。
该研究揭示了HRAS特异性m6A修饰位点与肿瘤进展之间的联系,这为靶向致癌Ras信号通路提供了一种新的策略。
文章索引
标题:Epitranscriptic regulation of HRAS by N6-methyladenosine drives tumor progression
发表期刊:Proc Natl Acad Sci U S A.
发表时间:2023.04
作者及单位:东南大学高山教授团队
IF:11.1
DOI:10.1073/pnas.2302291120.
研究思路

研究结果
(1)HRAS m6A修饰鉴定
与癌旁组织相比,HRAS的3'UTR在肿瘤组织中具有更高的m6A水平,而5'UTR和CDS没有明显的m6A修饰(图1A)。然而,在KRAS和NRAS中没有观察到明显的m6A修饰(图1A)。

m6A-RIP-qPCR分析发现:膀胱癌5637细胞系、肝癌HepG2细胞系(HRAS野生型)和膀胱癌T24细胞系(HRASG12V突变)的HRAS被m6A显著修饰(图1B)。此外,肾细胞癌组织中HRAS的m6A水平明显高于邻近组织(图1C)。
总之,这些数据表明,m6A修饰存在于HRAS中,而不存在于KRAS和NRAS中。

接着,确定HRAS m6A修饰的主要调控因子。m6A-RIP-qPCR分析显示,去甲基化酶FTO和ALKBH5敲低后,HRAS m6A修饰增强(图1D);野生型FTO和ALKBH5过表达,HRAS m6A修饰减弱(图1E)。

此外,RIP-qPCR显示FTO和ALKBH5与HRAS结合(图1F)。总之,这些结果证实了HRAS是FTO和ALKBH5催化的去甲基化的直接底物。

(2)m6A修饰可增强HRAS翻译
m6A修饰对HRAS表达存在哪些影响呢?
首先,荧光素酶报告基因(下图A)、亚细胞定位实验(下图B-C)和HXD处理(下图D-E)排除了m6A对HRAS转录、亚细胞定位、mRNA稳定性的调节。

qRT-PCR结果显示,FTO和ALKBH5 KD对HRAS、KRAS和NRAS的mRNA表达均无显著影响(图2A)。这些结果表明m6A对HRAS的RNA水平没有影响。

然而,FTO和ALKBH5 KD均显著增加了H-Ras蛋白水平(图2C-D)。相比之下,野生型FTO和ALKBH5的OE显著降低了H-Ras蛋白水平(图2E-F)。这些结果表明m6A可提高H-Ras蛋白水平。

这怎么回事??翻译组学来解释~~~
首先构建了pmirGLO-HRAS 3'UTR荧光素酶报告基因,探讨m6A是否调控HRAS的翻译。双荧光素酶实验显示,FTO和ALKBH5 KD后,荧光素酶活性显著增强,而萤火虫荧光素酶(F-Luc)和Renilla荧光素酶(R-Luc) mRNA丰度不受影响(图2H),表明去甲基酶调节了HRAS的翻译效率。

接下来,作者用两种方法进行核糖体捕获(23,24)。Polysome profiling及qRT-PCR显示,FTO和ALKBH5 KD后,HRAS主要分布在Polysome组分中(图2I)。

另外,RiboLace显示FTO和ALKBH5 KD后HRAS mRNA分布在活性核糖体结合的片段中。总之,这些数据表明m6A修饰与HRAS的翻译有关。

(3)YTHDF1以依赖m6a的方式促进HRAS翻译
RIP-qPCR检测显示,在5637和HepG2细胞中,YTHDF1与HRAS结合明显(图3A)。RIP-seq显示YTHDF1在HRAS的3'UTR中显著富集(图3B),证实了HRAS的3'UTR被YTHDF1特异性结合。

RIP- qPCR发现FTO和ALKBH5 KD后,YTHDF1与HRAS转录物的结合能力显著增强(图3C)。将YTHDF1的两个关键氨基酸突变(K395A和Y397A),以消除其与m6A的结合。Flag RIP-qPCR检测显示,HRAS与YTHDF1-wt的相互作用显著增强,但YTHDF1-mut与HRAS的相互作用明显减弱(图3D),提示YTHDF1与HRAS转录物的结合依赖于m6A。


Polysome profiling和RiboLace显示:YTHDF1 KD后,Polysome组分或翻译活性核糖体部分中的HRAS mRNA水平降低(图3F和G)。

YTHDF1促进m6A修饰基因的翻译,通常与翻译起始因子和延伸因子相关,如eIF3、eIF4、eEF1和eEF2。RIP-qPCR检测显示eEF1和eIF4与HRAS转录本显著结合(图3I)。FTO和ALKBH5 KD中eEF1对HRAS转录本的结合能力增强(图3 J和K),证实m6A通过YTHDF1和eEF1诱导HRAS mRNA的翻译延伸。
 综上所述,这些数据表明YTHDF1以依赖于m6A的方式促进了HRAS的翻译效率。
综上所述,这些数据表明YTHDF1以依赖于m6A的方式促进了HRAS的翻译效率。
- HRAS m6A修饰位点的鉴定
生物素化和m6A修饰的RNA探针pull-down实验进一步验证了YTHDF1与三个m6A位点的结合(图4E)。
最后,再综合m6A位点点突变的荧光素酶报告基因(图4F),以及HRAS-Mut和YTHDF1-Mut的IB分析(图4G)结果,表明HRAS 3' UTR包含三个m6A位点,这是YTHDF1结合和HRAS翻译所必需的。

- dm6ACRISPR消除HRAS m6A修饰,可抑制肿瘤生长


- FTO/YTHDF1/H-Ras 轴调控肿瘤生长

研究结论
该研究解析了HRAS的表观转录调控在驱动肿瘤发生发展中的重要作用与机制,为靶向Ras的肿瘤精准治疗提供了新的理论基础和策略。
欢迎致电了解详情或咨询!
广州卿泽生物科技有限公司
地址:广州市黄埔区伴河路96号一栋三层321房
电话:18925086102(微信同号)